

2025年7月,国际机器学习会议(InternationalConfer-ence on Machine Learning,ICML)将在加拿大温哥华举办。届时,来自全球的顶尖高校、研究机构、企业的研究者以及英伟达在内的诸多顶尖研究团队将在现场展示最新成果。
AI蛋白质设计公司「力文所」提交的科研论文《P(all-atom) ls Unlocking New Path ForProtein Design》成功入选 ICML,并获得“聚光灯海报展示(Spotlight)”的机会。

据介绍,本届大会共收到 12,107 份投稿,最终录取率为 26.9%。其中,仅2.6%的论文获得“聚光灯展示”资格,代表高质量、高推荐度的研究成果。
而力文所是其中之一。这是新锐科研力量能在国际舞台上发出清晰而坚定的声音的表现,也是对力文所团队学术上的认可,更是对力文所在AI+蛋白质设计交叉创新领域持续探索的深度肯定。

在国际舞台讲述中国创新
科研论文《P(all-atom) ls Unlocking New Path ForProtein Design》这项研究是由力文所人工智能与蛋白质设计方向的算法开发团队合作完成,凝聚了多位成员在模型设计、实验验证和论文撰写中的集体智慧。
以下为该项研究的主要内容:
目前,应用扩散模型进行蛋白质骨架生成已经非常成熟。但只生成骨架结构,还无法获得蛋白质序列,需要借助Inverse folding model预测蛋白质序列。这种“两步法”的方式存在不少缺陷,例如无法体现侧链对结构的影响,结构设计性受序列预测模型的影响等等。
为了解决上述问题,我们研发了Pallatom模型,它可以直接生成蛋白质的所有原子(all-atom),通过氨基酸侧链,可以自然地得到蛋白质序列信息,实现了端到端的全原子生成模型。
蛋白质全原子结构包括了骨架原子和侧链原子,骨架原子的类型和数量是确定的,但侧链原子会随着氨基酸类型的改变而发生明显变化。为了解决这一问题,模型引入了Atom14系统,为每个氨基酸定义标准的Atom14结构,不存在的原子,通过虚拟原子进行补足。由Pallatom生成的全原子结构最终可以在Atom14系统中激活对应的氨基酸类型,从而直接得到蛋白质序列。这就避免了序列和结构生成中“先有鸡还是先有蛋”的问题,因为侧链一旦确定氨基酸类型就确定了。
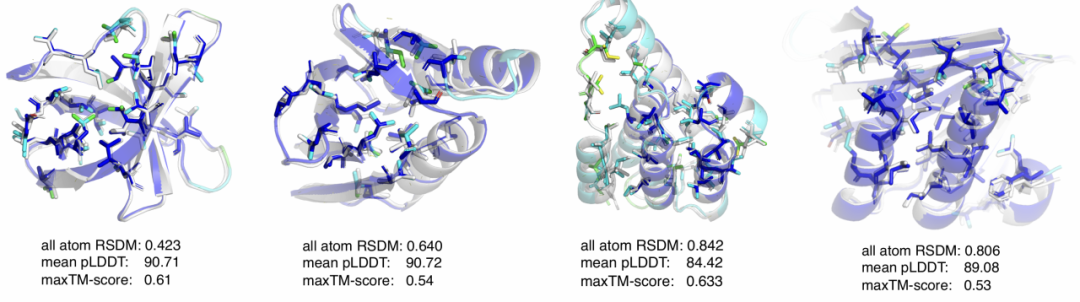
通过实验发现,Pallatom模型在蛋白质结构的可设计性、多样性和新颖性上都有非常强劲的表现,效果显著超越先前方法。此外,模型生成的全原子结构更加紧密和稳定,蛋白质的疏水侧链通过内聚作用,形成了稳定的疏水核心,能够维持蛋白质整体结构的稳定性。而表面的亲水极性残基又能与周围水分子相互作用,提高蛋白质在水溶液中的溶解性。模型先进的表现能力让我们非常惊喜,这为蛋白质设计开辟了新的道路。
关于国际机器学习会议(ICML)
ICML是人工智能领域最具影响力的国际顶会之一。它汇聚了全球最前沿的研究成果与思想,是AI学术界与产业界的重要风向标。
ICML 以展示和发表机器学习各个层面的前沿研究而闻名,涵盖人工智能、统计学、数据科学等基础学科,同时延伸至图像识别、计算生物学、语音理解、机器人技术等多个关键应用领域。